本記事では分子データをBlender上で扱い、可視化することを可能とするアドオンである「Molecular Nodes」の使い方を紹介します。
Molecular Nodesは分子データのインポート、アニメーション化、および操作をGeometry Nodes上で行うことができます。
Molecular NodesアドオンはBlender3.2 以上でのみ動作しますので、本記事ではBlender3.3LTSを使用します。
本アドオンでは一部機能を使用するためPython関連の操作がありますが、決して難しいものではないのでぜひチャレンジしてみてください。
導入方法
アドオン本体のインストール
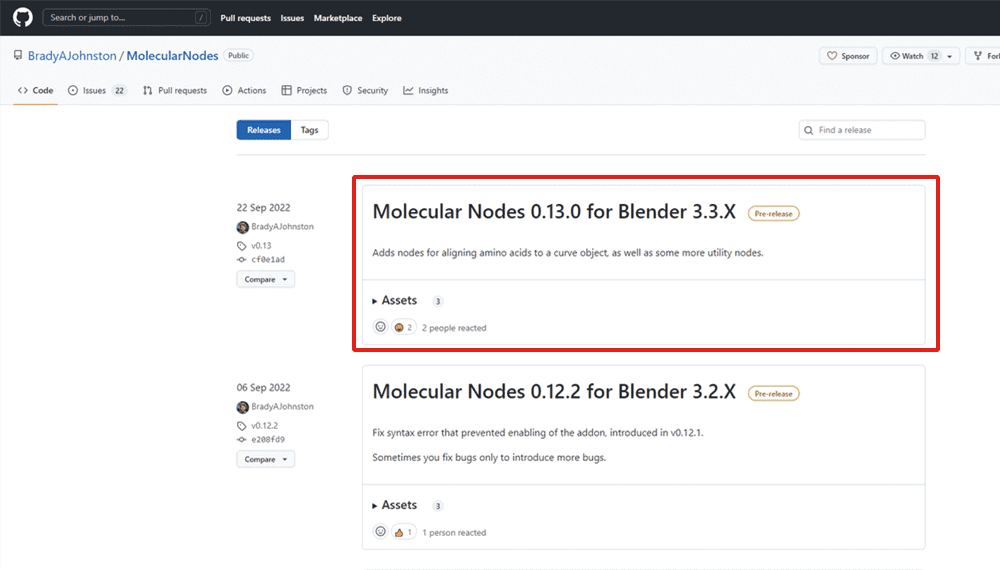
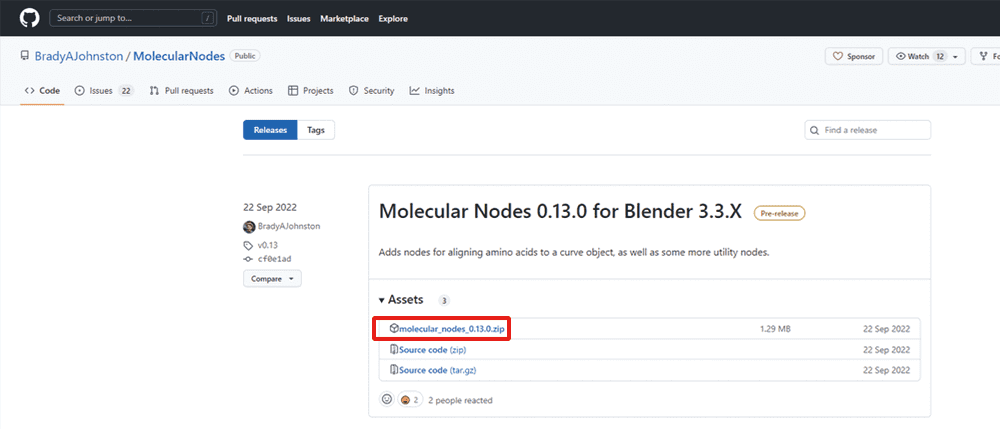
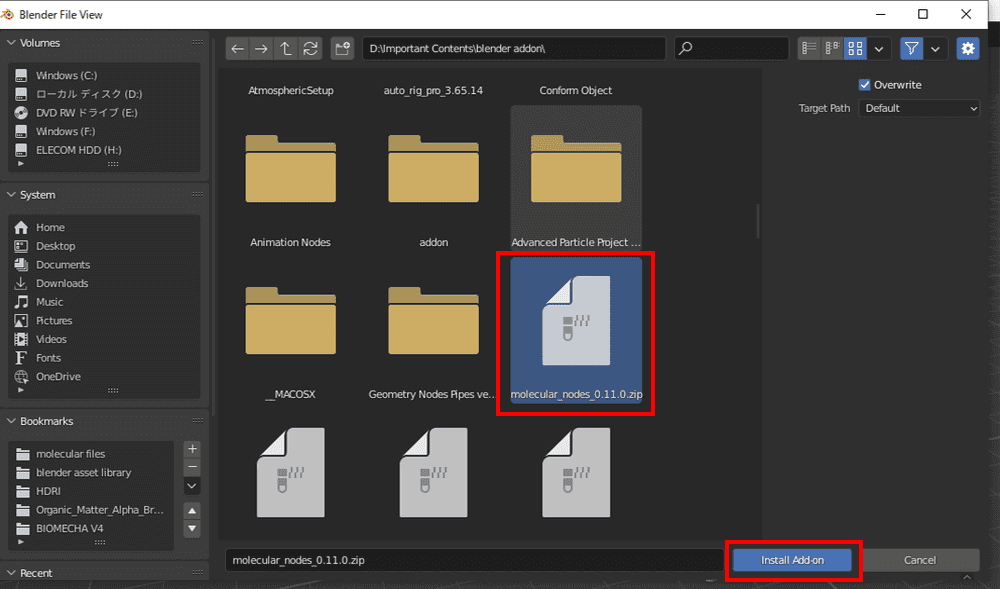
下記のURLからGithubにアクセスし、最新リリースのzipファイルをダウンロードします。
ダウンロードしたzipファイルを解凍せずに任意の場所に保存して下さい。
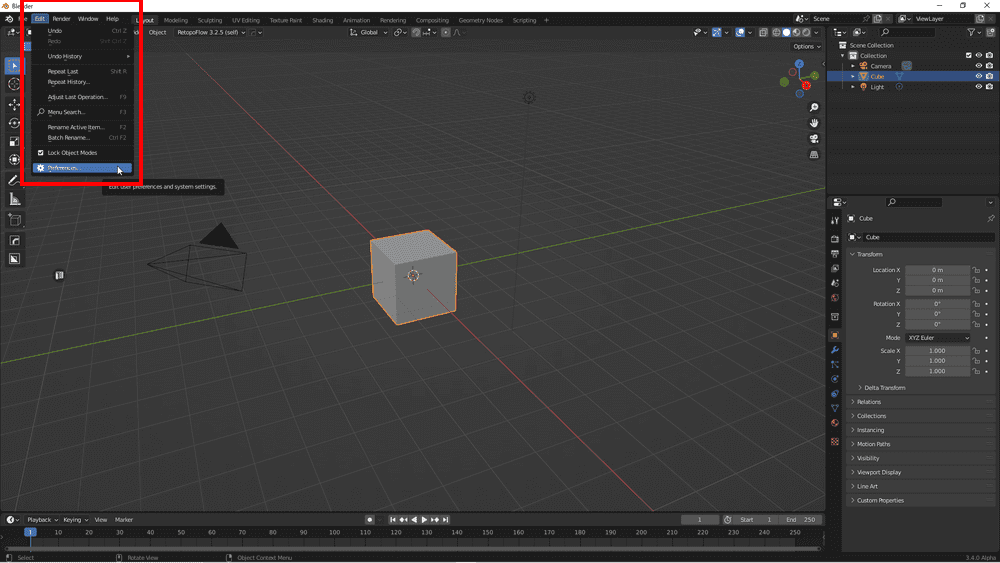
Blenderを起動し、Edit→PreferenceからAdd- onsを選択します。
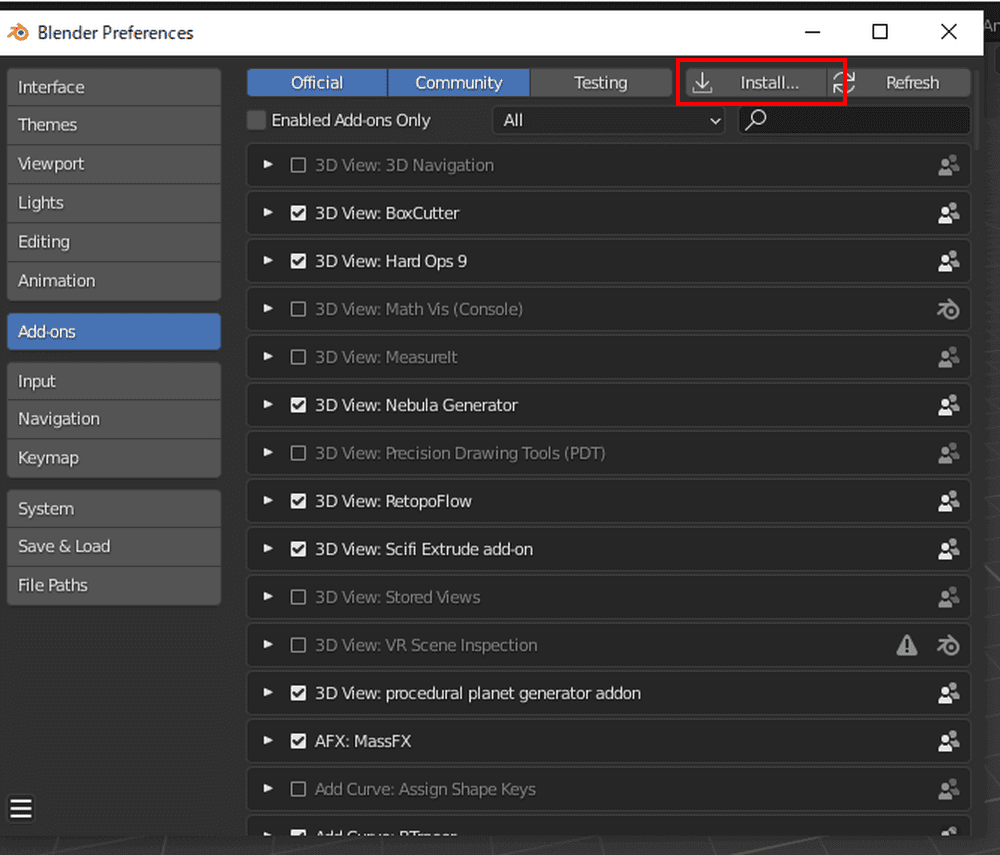
右上のInstallをクリックし、先ほど保存したzipファイルを選択しインストールします。
アドオンが表示されたら名前の横のボックスにチェックを入れて有効にします。
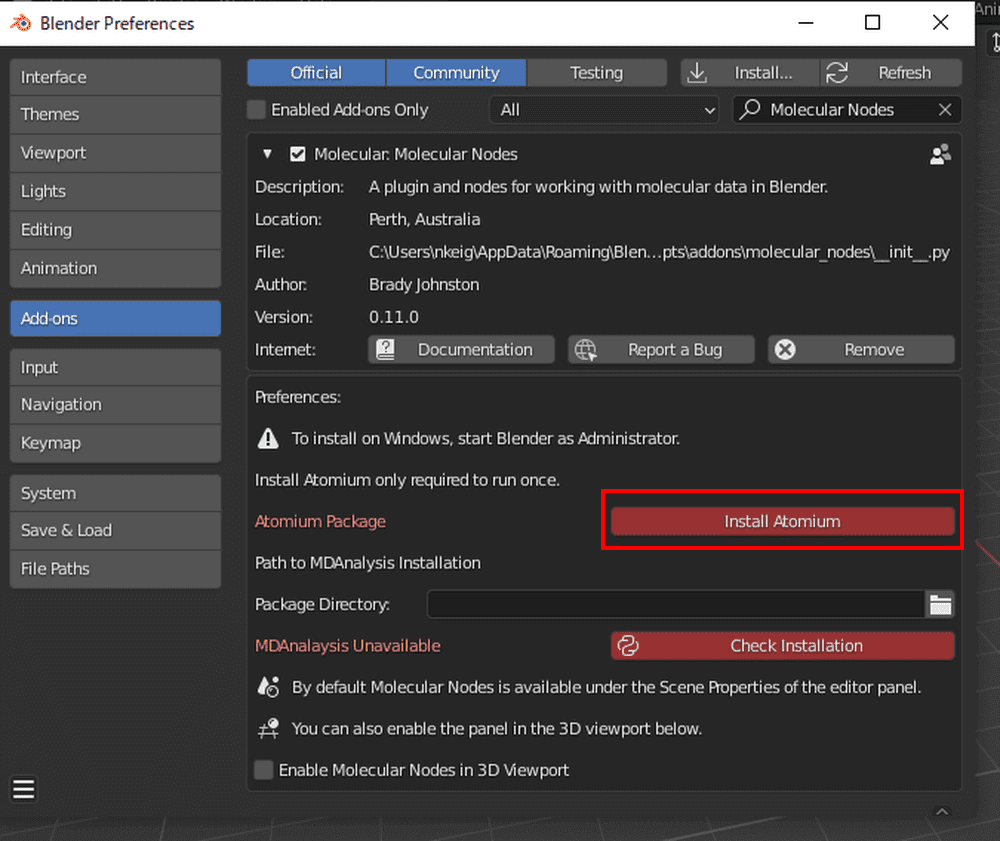
チェックボックスの横の三角マークをクリックして詳細を表示させます。
赤く表示されているInstall Atomiumをクリックしてください。
しばらく待つと色がグレーに変化したらAtomiumのインストール完了です。
MDAnalysisのインストール
複雑な分子データや、特殊な拡張子のデータを読み込むためにMDAnalysisをインストールし、Molecular Nodesアドオンと紐付けます。
アドオンの一部機能のみを使用する場合はMDAnalysisのインストールは必要ないので、余裕のある方のみ試してみることを推奨します。
Blender3.1以上はPython3.10以上にバンドルされているのでPython3.10以上をインストールします。
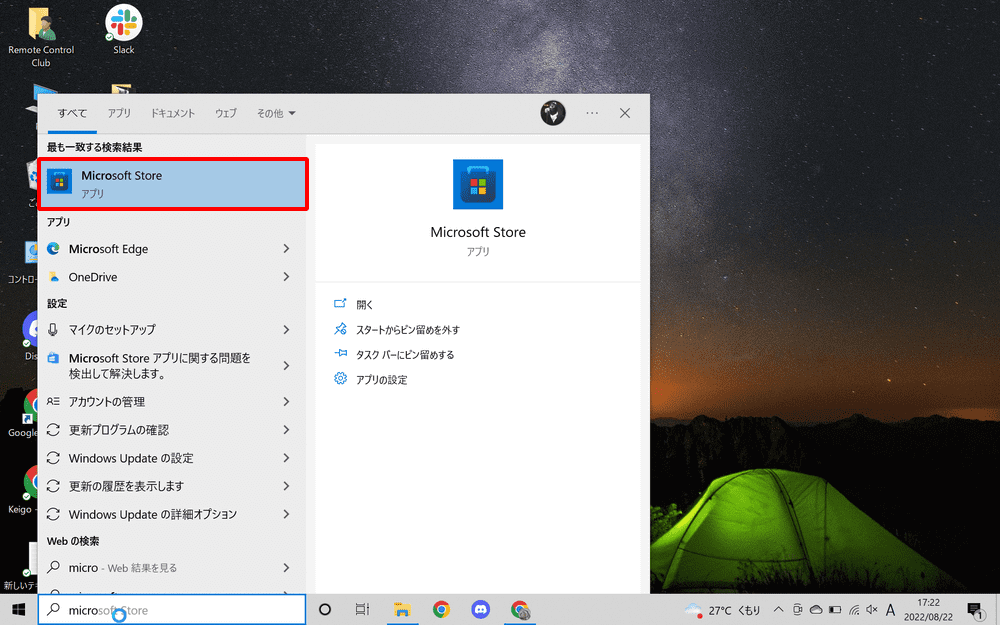
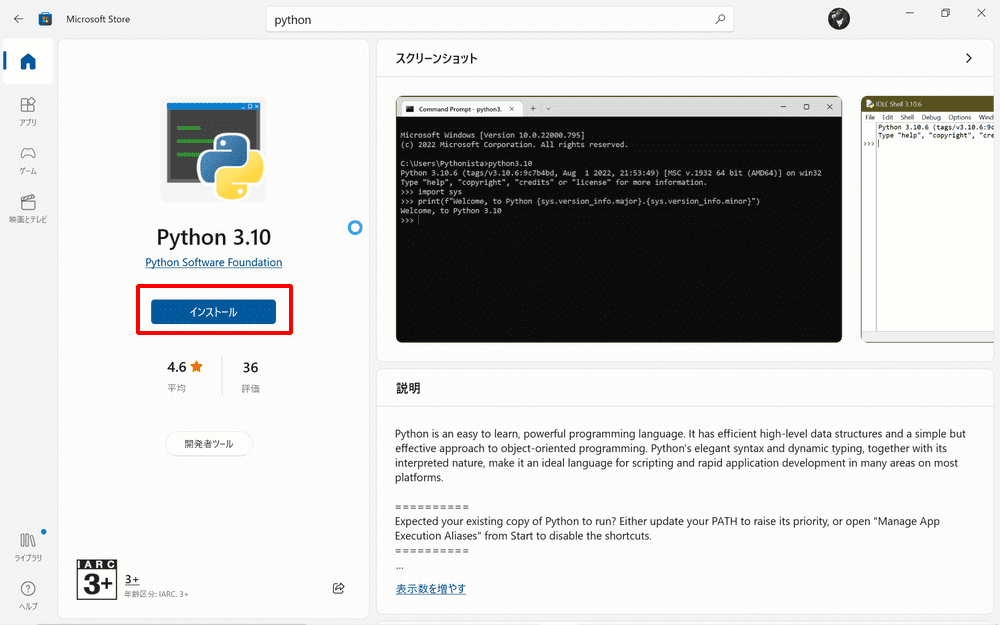
タスクバーの検索欄に「Microsoft Store」と入力し、起動します。
Microsoft StoreでPythonと検索し、Python3.10をインストールします。
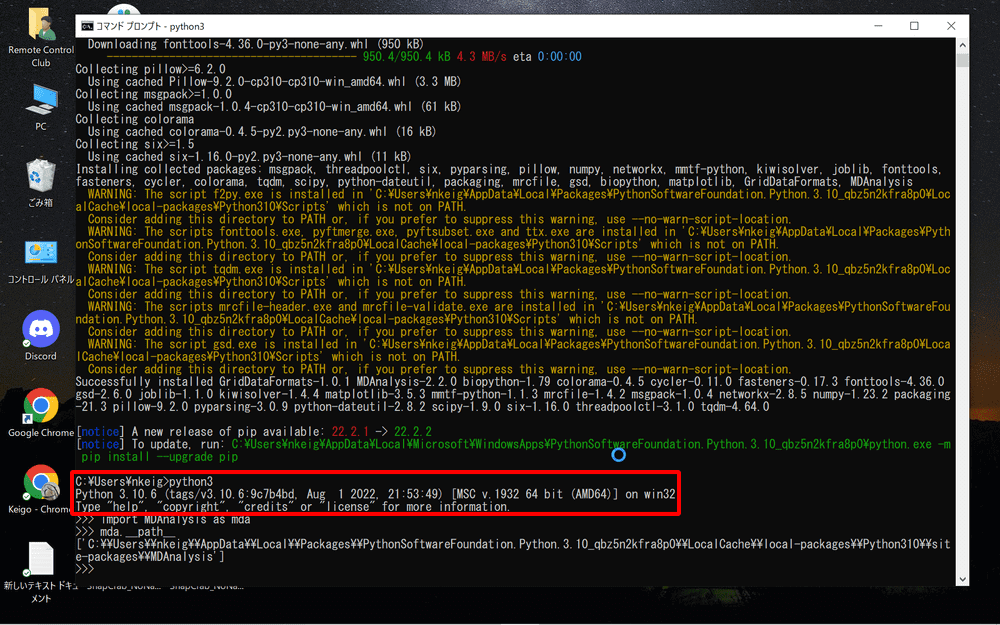
Pythonのダウンロードが完了したら、コマンドプロンプトを開きます。
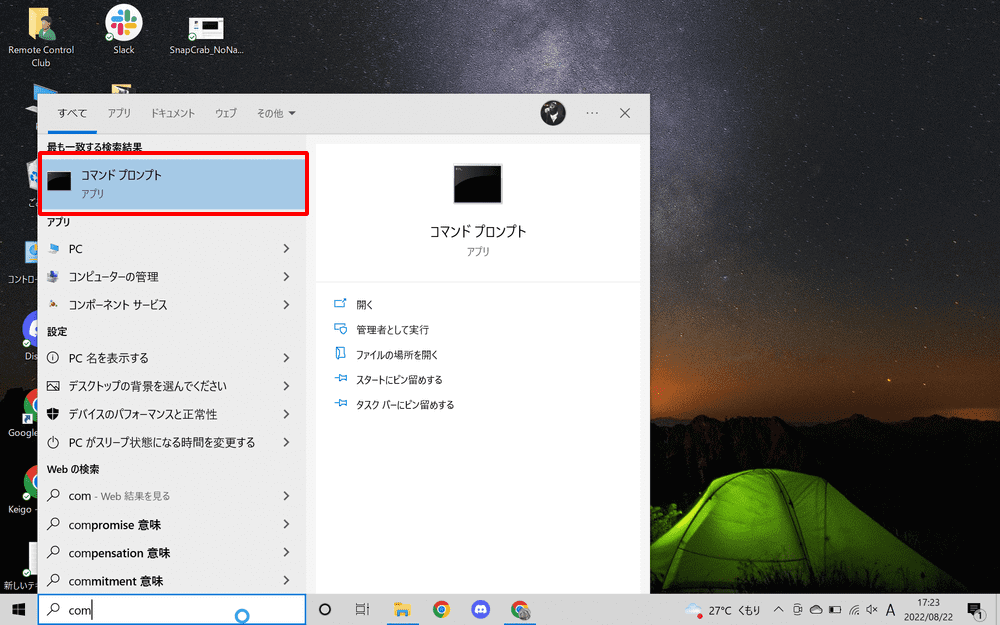
タスクバーの検索欄に「コマンドプロンプト」と入力し、起動します。
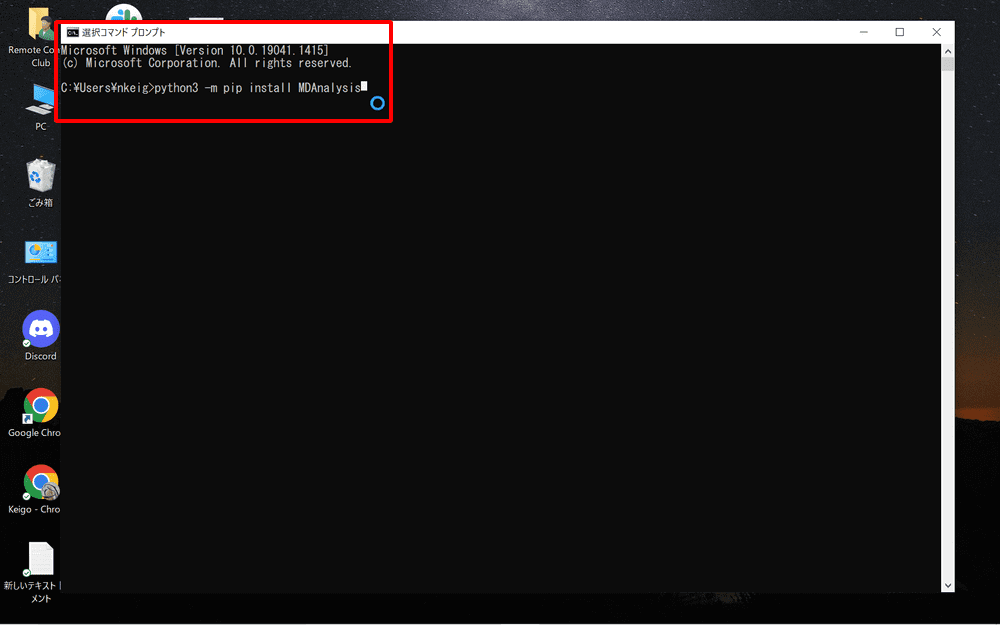
コマンドプロンプトを起動したら
python3 -m pip install MDAnalysis
をコピーして貼り付けます。
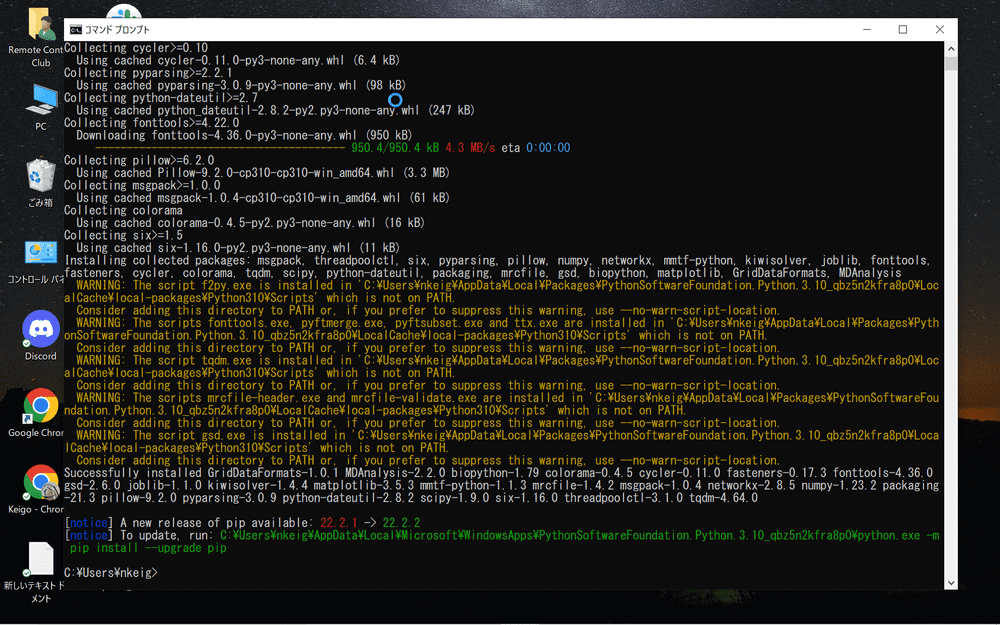
するとMDAnalysisがインストールされます。
インストールが完了したら、
python3
と入力してPythonを起動します。
次に
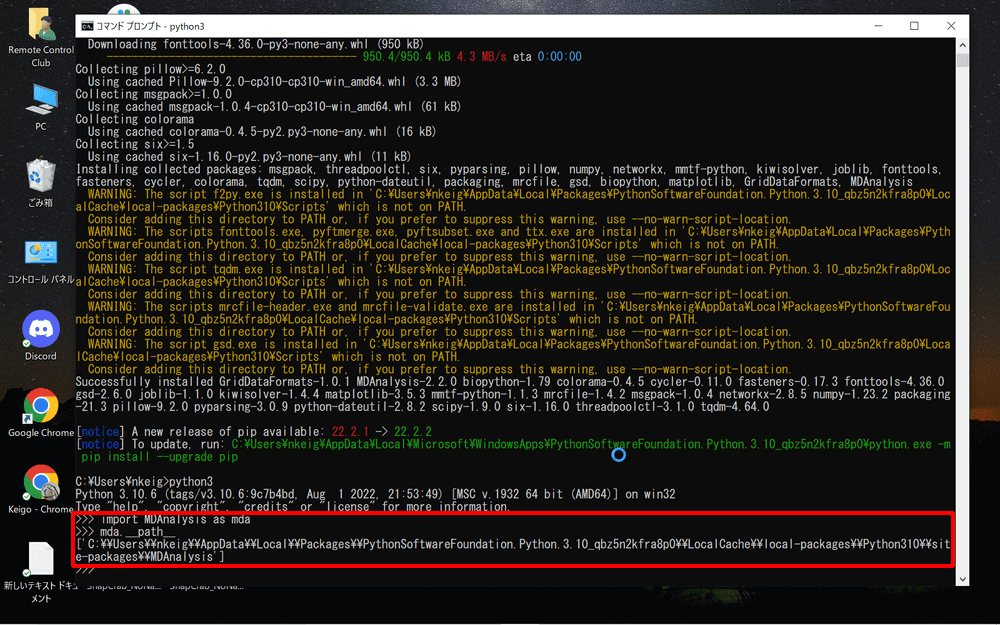
import MDAnalysis as mda
mda.__path__
と入力すると、MDAnalysisのパスが表示されます。
パスのC~からsite packagesまでをコピーし、Blenderに戻ります。
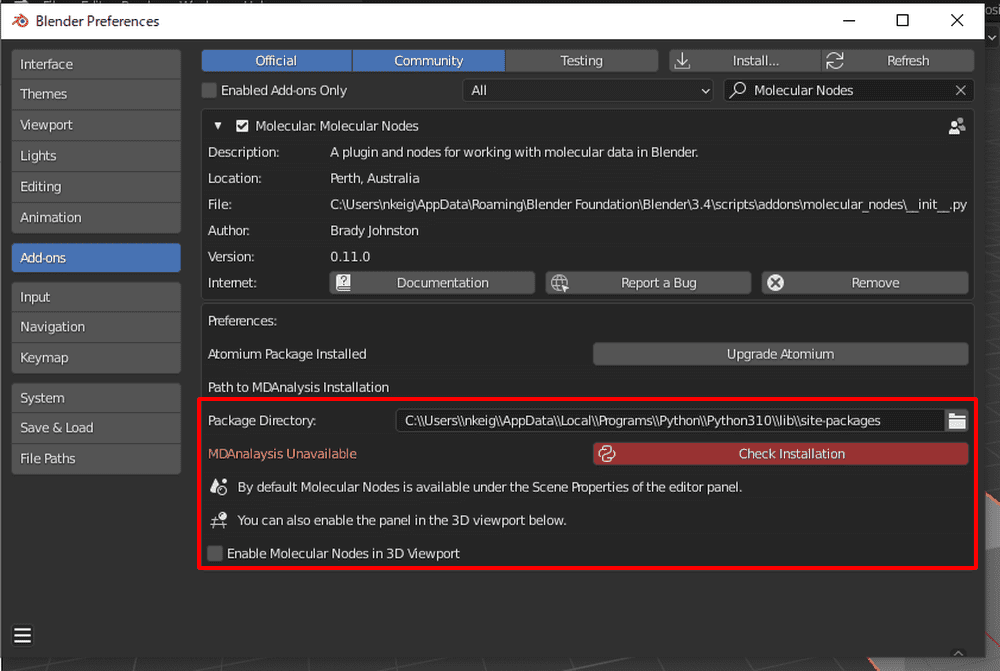
私の場合は「C:\\Users\\nkeig\\AppData\\Local\\Programs\\Python\\Python310\\lib\\site-packages」といった様になります。
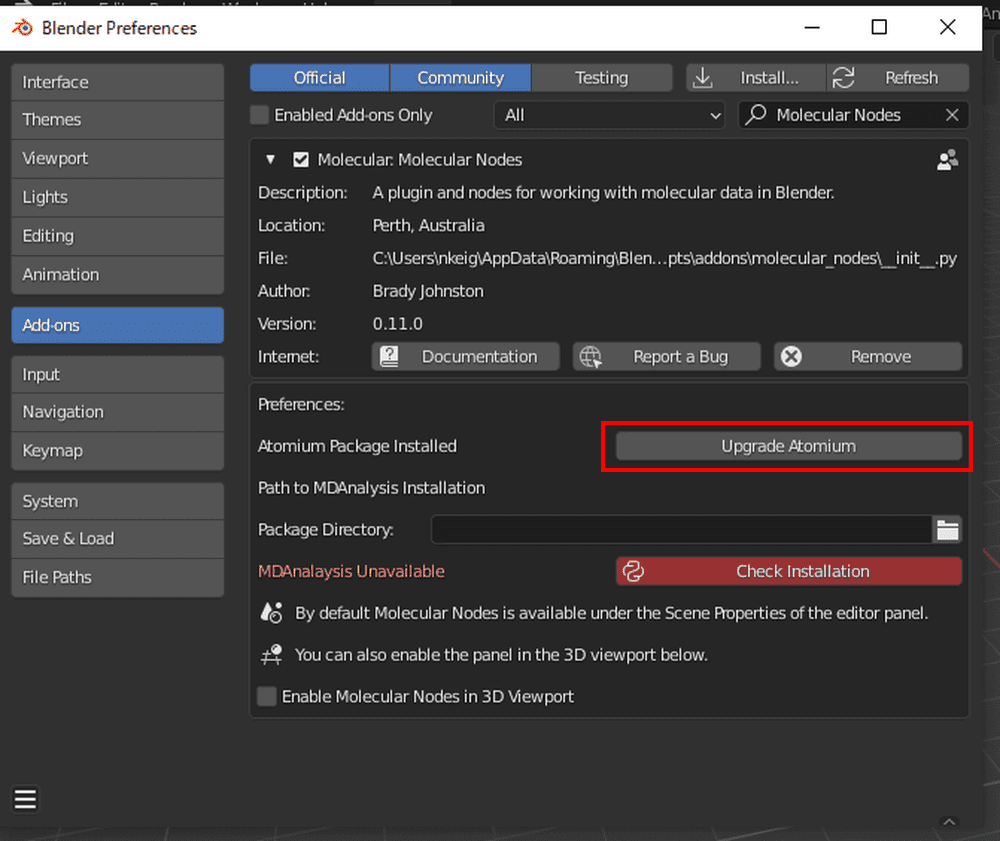
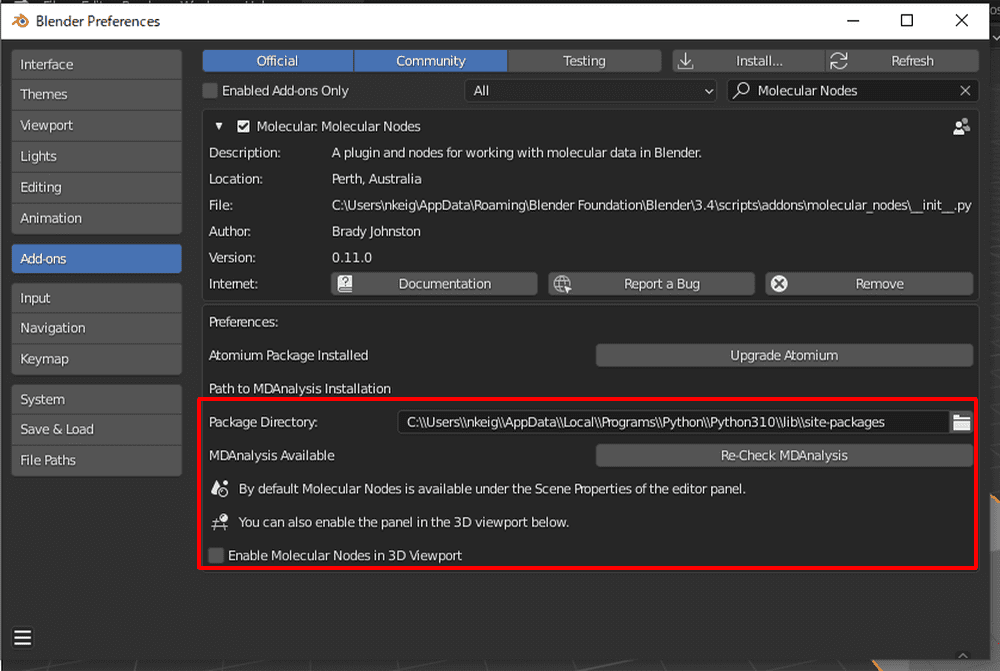
Blenderに戻ったらMolecular NodesアドオンのPackage Directoryにパスを貼り付け、Check Installationをクリックします。
しばらくして色がグレーになったら成功です。
これでMolecular Nodesの全てのインストールは完了です。
Molecular Nodes Addonの具体的な使用方法は以下の記事で詳しく解説しています。
STYLYに関する質問、バグ報告、改善してほしい要望はSTYLY FORUMまで
https://jp.forum.styly.cc/support/discussions
Certified (QA) by Shota Shawn Yoshizawa
Edited by SASAnishiki